User:Gaston Paris:Notebook/Mutantes CheY
Objetivo
Construir las cepas mutantes de CheY, BAB1_0100 en B. abortus 2308 y clonar los genes correspondientes en pTrcHis. Evaluar la infección en macrófagos en condiciones de luz y oscuridad.
Estrategia
Mutantes
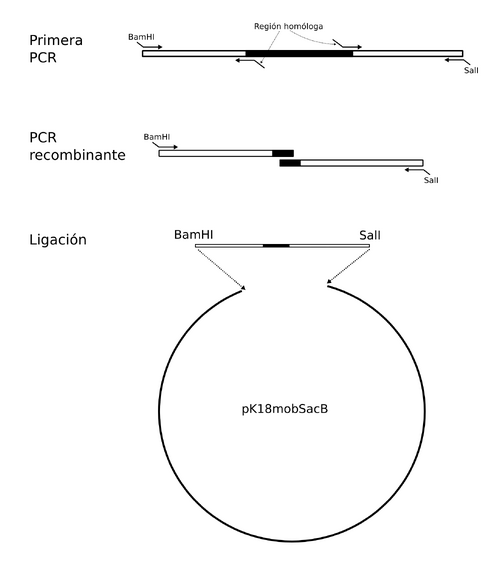
Se amplificaran las regiones 5' y 3' de cada gen para luego purificarlas y realizar una PCR recombinante con los fragmentos. El fragmento recombinante se digerirá y purificará por gel para luego clonarlo en el vector pK18mobSacB.
Se utilizarán los siguientes oligos:
5CheyFWD-BamHI y 5CheyREV fragmento 5' de CheY
3CheyFWD y 3CheyREV-SalI fragmento 3' de CheY
5Ba_0100FWD-BamHI y 5BAB_0100REV fragmento 5' de BAB1_0100
3BAB_0100FWD y 3BAB_0100REV-SalI fragmento 3' de BAB1_0100
La presencia del inserto correcto se verificará por colony PCR y digestión.
Los clones adecuados se transformaran en E.coli S-17 para luego movilizarlos a Brucella abortus 2308 por conjugación. Los clones simple recombinantes se seleccionaran por resistencia a Kanamicina, Ampicilina y ácido nalidíxico.
Los dobles recombinantes se seleccionaran por crecimiento en presencia de sacarosa.
El knock-out se confirmará por colony PCR y secuencia
Clonado Proteínas
Se amplificaran los genes CheY de B. abortus (BAB1_0099) y el regulador transcripcional río arriba de él (BAB1_0100). Los fragmentos amplificados se digeriran y purificaran por gel de agarosa para luego ligarlos al vector pTrcHisB (Invitrogen)
Se utilizaran los siguientes oligos:
CheY_FWD_Nhe y BAB1_0099_reverse para el gen CheY.
BAB_0100FWD_Nhe y BAB_0100REV_XhoI para el regulador transcripcional.
Resultados
Martes 30/10/07
Transformé 80 μl de DH5α con 2 μl de pK18mobSacB que me enviaron de Japón. Luego de recuperar en SOC, se inocularon 25 ml de SOB, que se pusieron a crecer a 37°C O.N.
Miércoles 31/10/07
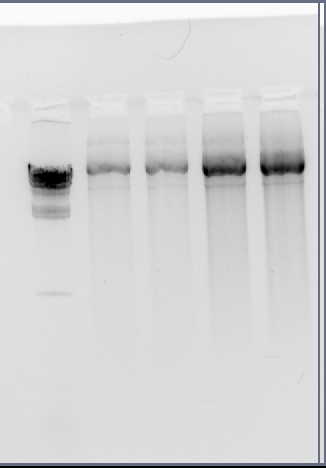
Se cosecharon 6 ml de cultivo de pK18monSacB por duplicado y se realizaron minipreps con el kit Qiagen utilizando el doble de volumen indicado en el protocolo estándar. Se corrieron alícuotas en un gel de agarosa para confirmar la cantidad y calidad. Se guardan a -20°C.
Jueves 1/11/07
Llegaron los oligos. Los diluyo a 10 μM y preparo el protocolo para amplificar.
Viernes 2/11/07
Prepare reacciones de PCR de acuerdo a la siguiente receta.
| Primer FWD | 5 μl |
| Primer REV | 5 μl |
| gDNA Brucella 100 ng/μl | 1 μl |
| 10X Buffer pfu | 7,5 μl |
| dNTPS 2 mM | 5 μl |
| pfu FIL | 1 μl |
| H2O | 25,5 μl |
A cada tubo se agregaron los oligos y luego 33,7 μl de la siguiente mix:
| gDNA Brucella 100 ng/μl | 6,5 μl |
| 10X Buffer pfu | 48,75 μl |
| dNTPS 2 mM | 32,5 μl |
| pfu FIL | 1 μl por tubo |
| H2O | 165,75 μl |
Las condiciones de ciclado en la PCR Genius fueron:
- 5' a 94°C
- Hot Start a 80°C (se agregó 1 μl de pfu a cada tubo)
- Seguido de 35 ciclos de:
- 30" a 93°C
- 1' a 55°C
- 1' a 68°C
- Y 10' a 72°C de extensión final
Lo siguiente es la referencia:
| Tubo Nro | Primer FWD | Primer REV |
|---|---|---|
| 1 | 5CheyFWD-BamHI | 5CheyREV |
| 2 | 3CheyFWD | 3CheyREV-SalI |
| 3 | 5Ba_0100FWD-BamHI | 5BAB_0100REV |
| 4 | 3BAB_0100FWD | 3BAB_0100REV-SalI |
| 5 | CheY_FWD_Nhe | BAB1_0099_reverse |
| 6 | BAB_0100FWD_Nhe | BAB_0100REV_XhoI |
Lunes 5/11/07
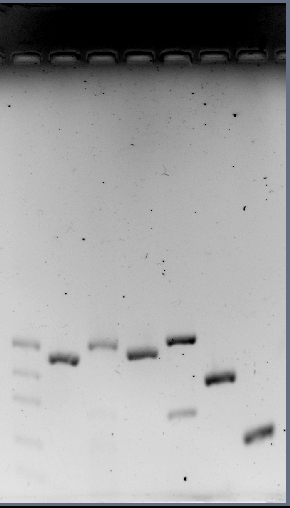
Se corrió un gel con 20 μl de cada PCR 1 a 6 y se cortaron las bandas correspondientes
Martes 6/11/07
Las bandas cortadas ayer se purificaron por Illustra GFX PCR (GE). Se guardan congeladas hasta mañana.
Miércoles 7/11/07
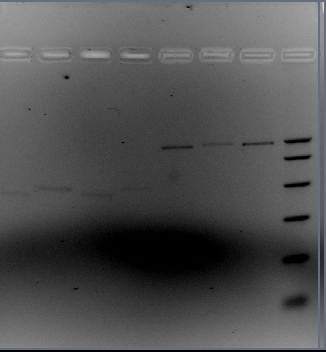
Corrí un gel con 5 μl de cada purificación.
Jueves 08/11/07
PCR recombinante de armado de la estrategia de deleción de CheY y BAB1_0100.
| CheY | BAB1_0100 | |||
|---|---|---|---|---|
| 5 CheYFWD BamHI | 5 μl | 5 Ba_0100FW BamHI | 5 μl | |
| 3 CheYREV SalI | 5 μl | 3 BAB_0100REV SalI | 5 μl | |
| Frag PCR1 | 10 μl | Frag PCR3 | 10 μl | |
| Frag PCR2 | 10 μl | Frag PCR4 | 10 μl | |
| 10X Buffer Pfu | 15 μl | 10X Buffer Pfu | 15 μl | |
| dNTPs 2mM | 10 μl | dNTPs 2mM | 10 μl | |
| pfu FIL | 1 μl | pfu FIL | 1 μl | |
| H20 | 44 μl | H20 | 44 μl |
Las condiciones de la PCR genius fueron las siguientes:
- 5' a 94°C
- Hot Start a 80°C (se agregó 1 μl de pfu a cada tubo)
- Seguido de 35 ciclos de:
- 30" a 93°C
- 1' a 55°C
- 2' a 68°C
- y 10' a 72°C de extensión final
Las PCR 5 y 6 del 7/11/07 se extrajeron una vez con cloroformo y se precipitaron con 0,7 volumenes de isopropanol y 0,3M de acetato de sodio.
Viernes 09/11/07
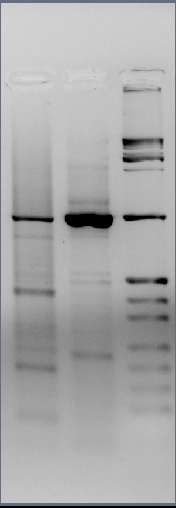
Corrí un gel con 10 μl de la PCR recombinante. Aparecen las bandas esperadas de 1 Kb.
Extraigo una vez con cloroformo y precipito con isopropanol + 0,3 M acetato de sodio.
Digestiones de los fragmentos precipitados:
- Para clonar los genes Chey y BAB_0100 con NheI-XhoI, junto con el vector pTrcHisB
- Para clonar los fragmentos recombinantes con BamHI-SalI, junto con el vector pK18mobSacB
| CheY Recombinante | BAB_0100 Recombinante | pK18mobSacB | |
|---|---|---|---|
| H20 | 18 μl | 18 μl | DNA miniprep 18 μl |
| NEBuffer 3 10X | 2,5 μl | 2,5 μ | 2,5 μ |
| BSA 10X | 2,5 μl | 2,5 μl | 2,5 μl |
| BamHI | 1 μl | 1 μl | 1 μl |
| SalI (NEB) | 1 μl | 1 μl | 1 μl |
| Volumen final | 25 μl | 25 μl | 25 μl |
| Gen CheY | Gen BAB_0100 | pTrcHisB | |
|---|---|---|---|
| H20 | 18 μl | 18 μl | DNA QIAGEN 18 μl |
| NEBuffer 2 10X | 2,5 μl | 2,5 μ | 2,5 μ |
| BSA 10X | 2,5 μl | 2,5 μl | 2,5 μl |
| NheI (NEB) | 1 μl | 1 μl | 1 μl |
| XhoI (NEB) | 1 μl | 1 μl | 1 μl |
| Volumen final | 25 μl | 25 μl | 25 μl |
Se incubó a 37°C 2-3hs. Luego se congelaron.
Lunes 12/11/07
Corrí un gel con las digestiones del viernes y las PCR de CheY y BAB1_0100.
Corté las bandas y las purifiqué por Qiaquick.
Martes 13/11/07
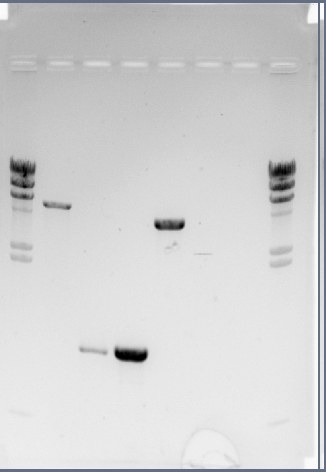
Corrí 3 μl de cada purificación en un gel de agarosa. Cuantificación:
- pK18mobSacB 20 ng/μL
- ΔCheY 10 ng/μL
- ΔBAB_0100 80 ng/μL
- pTHB 50 ng/μL
- CheY y BAB_0100 considero 10 ng/μL porque se fueron del gel.
Ligación
| Control - | ΔCheY | ΔBAB_0100 | |
|---|---|---|---|
| pK18mobSacB | 1 μL | 1 μL | 1 μL |
| ΔChey | - | 1 μL | - |
| ΔBAB_0100 | - | - | 1 μL |
| Buffer ligasa 10X | 1 μL | 1 μL | 1 μL |
| Ligasa 1/10 | 1 μL | 1 μL | 1 μL |
| H2O | 7 μL | 6 μL | 6 μL |
| Control - | CheY | BAB_0100 | |
|---|---|---|---|
| pTHB | 0,5 μL | 0,5 μL | 0,5 μL |
| Chey | - | 1 μL | - |
| BAB_0100 | - | - | 1 μL |
| Buffer ligasa 10X | 1 μL | 1 μL | 1 μL |
| Ligasa 1/10 | 1 μL | 1 μL | 1 μL |
| H2O | 7 μL | 6 μL | 6 μL |
Las ligaciones las guardé congeladas hasta que regreso de SAIB
Miércoles 27/11/07
Transformé 50 μL de células DH5α electrocompetentes con 5 μL de cada ligación. La recuperé con 1 mL de SOC y plaquee 200 μL en placas de LB ampicilina 100 ng/mL y kamicina 25 ng/mL.
Jueves 28/11/07
Las placas parecen estar correctas. El clonado en pTrcHisB funcionó correctamente y en pK18mobSacB está en el mismo nivel que el control
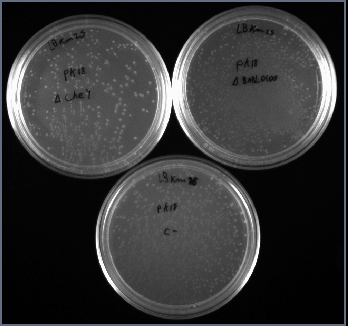
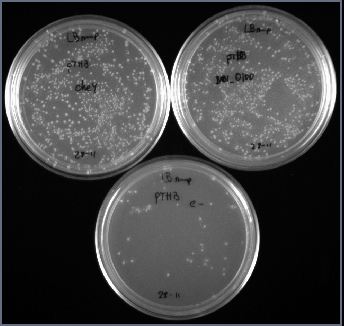
Lunes 03/12/07
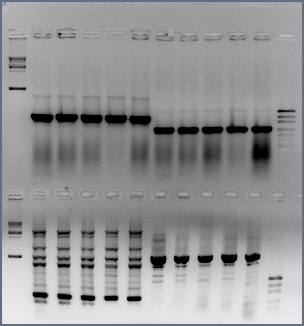
Corrí una colony PCR tomando 5 colonias de las placas de la ligación.
MIX
| X1 | X7 | |
|---|---|---|
| Primer FWD | 1 μL | 7 μL |
| Primer REV | 1 μL | 7 μL |
| 10X Buffer Taq | 2 μL | 14 μL |
| Cl2Mg 50 mM | 0,6 μL | 4,2 μL |
| dNTPS 2 mM | 2 μL | 14 μL |
| Taq FIL | 1 μL | 7 μL |
| H2O | 12,4 μL | 86,8 μL |
Se agregaron 20 μL a cada tubo en el cual se adicionó una colonia con un tip. El tipo se introdujo en 2 mL de LB con 100 ng/mL de ampicilina o 25 ng/mL de kanamicina y se incubó a 37°C
Las condiciones de la PCR genius fueron las siguientes:
- 5' a 94°C
- Seguido de 35 ciclos de:
- 30" a 93°C
- 30" a 55°C
- 45" a 72°C
- y 10' a 72°C de extensión final
Viernes 14/12/07
Comprobé las presencia de insertos en minipreps de los clones positivos. Solamente los clones en pTrcHisB con positivos y no los pK18mobSacB. Renombro los plásmidos: pTrcHisB-CheY a pGP0100 y pTrcHisB-BAB1_0100 a pGP0101.
Lunes 17/12/07
Retomo el clonado en pK18mobSacB. Voy a rehacer las PCRs recombinantes, posterior digestión y ligación.
Utilizo la enzima Pfx de Invitrogen para repetir la PCR recombinante. Comienzo con los fragmentos amplificados y purificados del 2/11/07 nombrado PCR1, 2, 3 y 4.
Protocolo PCR
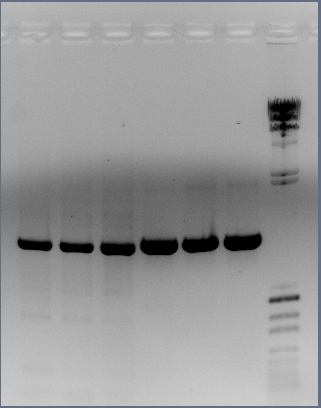
| C1 | C2 | C3 | |
|---|---|---|---|
| 5CheyFWD-BamHI | 1 μL | 1 μL | 1 μL |
| 3CheyREV-SalI | 1 μL | 1 μL | 1 μL |
| PCR1 | 5 μL | 5 μL | 5 μL |
| PCR2 | 5 μL | 5 μL | 5 μL |
| 10X Amplification Buffer (Invitrogen) | 10 μL | 10 μL | 10 μL |
| 50 mM SO4Mg | 1 μL | 1 μL | 1 μL |
| 2 mM dNTPs | 7,5 μL | 7,5 μL | 7,5 μL |
| 10X PCR Enhancer Solution (Invitrogen) | - | 5 μL | 10 μL |
| Pfx polymerase (Invitrogen) | 0,5 μL | 0,5 μL | 0,5 μL |
| H2O | 19 μL | 14 μL | 9 μL |
| B1 | B2 | B3 | |
|---|---|---|---|
| 5Ba_0100FWD-BamHI | 1 μL | 1 μL | 1 μL |
| 3BAB_0100REV-SalI | 1 μL | 1 μL | 1 μL |
| PCR3 | 5 μL | 5 μL | 5 μL |
| PCR4 | 5 μL | 5 μL | 5 μL |
| 10X Amplification Buffer (Invitrogen) | 10 μL | 10 μL | 10 μL |
| 50 mM SO4Mg | 1 μL | 1 μL | 1 μL |
| 2 mM dNTPs | 7,5 μL | 7,5 μL | 7,5 μL |
| 10X PCR Enhancer Solution (Invitrogen) | - | 5 μL | 10 μL |
| Pfx polymerase (Invitrogen) | 0,5 μL | 0,5 μL | 0,5 μL |
| H2O | 19 μL | 14 μL | 9 μL |
Las condiciones en la PCR Genius fueron las siguientes:
- 5' a 94°C
- Hot Start a 80°C
35 ciclos de:
- 30" a 93°C
- 1' a 55°C
- 1' a 68°C
seguido de un ciclo:
- 10' a 68°C
Precipitación
A las reacciones les agregué 150 μL de H2O y realicé una extracción con cloroformo. Centrifugué 5 min a máxima velocidad y tome la fase superior, a la cual le adicioné 0,1 volúmenes de AcNa 3M y 0,7 volúmenes de Isopropanol. Centrifugué 25 min a máxima velocidad. Lavé el pellet con etanol 70% y deje secar.
Digestión
Repetí las digestiones como el 9/11/07. Incluí dos tubos control con el plásmido pTrcHisB
| pTrcHisB (Qiagen) | 3 μL | 3 μL |
| NeBuffer 3 10X | 2,5 μL | 2,5 μL |
| BSA 10X | 2,5 μL | 2,5 μL |
| BamHI | 1 μL | - |
| SalI | - | 1 μL |
| H2O | 17 μL | 17 μL |
1 h 37°C
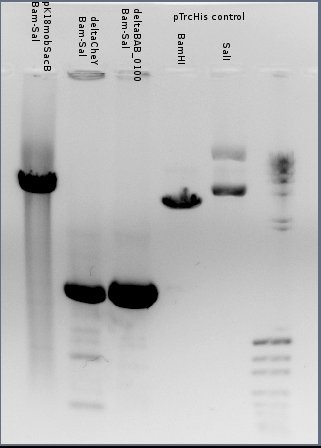
Martes 18/12/07
Corro un gel con las digestiones de ayer y purifico las bandas de pK18, ΔCheY y ΔBAB1_0100 por Qiaquick.