BIOMOD/2012/Team Sendai/Results & Discussion/AFM
<html xmlns="http://www.w3.org/1999/xhtml"> <head> <meta http-equiv="Content-Type" content="text/html;charset=utf-8" /> <title>SAMPLE</title> <style type="text/css">
a {color: #017acd}
/* コンテナ */ div#Container {background-image: url(side-blue.png); background-repeat: repeat-y; background-position: right top; width: 800px; margin-left: auto; margin-right: auto}
/* ヘッダー */ div#Header {background-color: #ffffff; background-image: url(sky.jpg); padding: 28px 20px 50px}
div#Header h1 {margin: 0}
div#Header p {color: #000000; font-size: 1.5em; font-family: Times New Roman, Times, serif; margin: 0}
/* パンくずリスト */ p#Bread {background-color: #ffffff; font-size: 1em; margin-top: 0; margin-bottom: 0; padding-top: 3px; padding-bottom: 20px}
/* コンテンツ */
div#Content {width: 580px;
float: left;
margin-left: auto;
margin-right: auto;
margin-bottom: 25px}
div#Content h2 {background-color: #808080; background-image: url(bar-blue.png); background-repeat: no-repeat; font-size: 1.25em; font-family: Lucida Sans Unicode, Courier, sans-serif; color: #ffffff; line-height: 28px; padding-left: 6px; margin-top: 0; margin-bottom: 0}
div#Content h3 {background-color: #e0e0e0; background-image: url(bar-green.png); background-repeat: no-repeat; font-size: 0.875em; line-height: 23px; padding-left: 30px; margin-top: 30px; margin-bottom: 0}
div#Content p {font-size: 1.5em; font-family: Times New Roman, Times, serif; line-height: 1.6; margin-top: 10px}
div#Content p#message {margin-top: 0}
/* サイドバー */
div#Sidebar {width: 190px;
float: right;
margin-bottom: 25px}
ul.sidemenu {border-top: solid 1px #000000; font-size: 1.25em; font-family: Times New Roman, Times, serif; margin-top: 20px; margin-left: 12px; margin-right: 12px; padding-left: 0; line-height: 0}
ul.sidemenu li {list-style-type: none}
ul.sidemenu li a {display: block; line-height: 30px; text-decoration: none; color: #000000; background-color: transparent; border-bottom: solid 1px #000000; padding-left: 5px}
ul.sidemenu li a:hover {background-color: #017acd}
p.feed {margin-bottom: 10px; margin-left: 12px;}
p.feed a {background-color: #ffffff; font-size: 0.75em; color: #444444; text-decoration: none; line-height: 30px; border: solid 1px #888888; padding: 5px}
p.feed img {border: none; vertical-align: middle}
/* フッター */
div#Footer {background-image: none;
background-repeat: repeat-x;
width: 190px;
margin-left: 570px;
margin-top: 0;
padding-top: 8px;
clear: both}
address {font-size: 0.75em; font-style: normal; color: #000000; text-align: left; padding: 12px}
</style> </head> <body>
<img src="http://openwetware.org/images/3/35/Formattop2.jpg " alt="Mother Cube2" "width="800" height="141"//>
<a href="http://openwetware.org/wiki/Biomod">BIOMOD</a> > <a href="http://openwetware.org/wiki/Biomod/2012 ">2012</a> > <a href="http://openwetware.org/wiki/Biomod/2012/Tohoku/Team_Sendai ">Team Sendai</a> > <a href="http://openwetware.org/wiki/Pilot_ver. ">Team Sendai A Top</a>> <a href=" http://openwetware.org/wiki/Methods.html ">Methods</a>
<a href="feed.xml"> <img src="feed-icon-14x14.png" alt="RSS" /> RSSフィード </a>
<実験>
STAGE1目的:脂質でDNAオリガミが引っ付くかの確認
①BIOMOD2011のフィールドおよび2D robotを製作する。
昨年BIOMODで用いたDNAorigamiのレースフィールドおよびロボット本体であった三角柱を作成する。
参考[1]
実験条件
| ステイプル(field)① | ステイプル(field)② |
|---|---|
| フィールドステイプル222本 各1μL | M13(84nM) 4.8μL |
| mQ 33μL | 10×TAE Mg2+ 10μL |
| ステイプル(field)① 10μL | |
| mQ 75.2μL | |
| 計 250μL | 計 100μL |
ステイプル(field)②を95°C~25°Cまで1°C2分の条件でサーマルサイクラーにかけ、アニーリングを行う。
| ステイプル(2D robot)① | ステイプル(2D robot)② |
|---|---|
| 2D robotステイプル33本 各1μL | M13(84nM) 10μL |
| mQ 92μL | 5×TAE Mg2+ 20μL |
| ステイプル(2D robot)① 10μL | |
| mQ 60μL | |
| 計 125μL | 計 100μL |
フィールドステイプルと同様、95°C~25°Cまで1°C2分の条件でサーマルサイクラーにかけ、アニーリングを行う。
これらを高速AFMにより観察した結果が以下Figure1.1 Figure1.2である。
 |
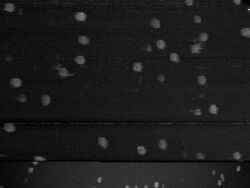 |
field DNA origamiはかなりの数が作成できていることが確認できた。しかしながら2D robotは複数の欠片が生成できただけで目的の形は観察されなかった。この欠片は丸い形をしており、多数のステイプルがアグリゲーションしていると思われる。
よって以下の実験はfield DNA origami を用いて進める。
②DNAオリガミの精製を行う。(PEG沈、エタ沈)
Field DNA origamiの作成はできたが、溶液中には余分なステイプルが多々入っており、これらが脂質を加えた際、悪影響を及ぼす可能性も挙げられる。
ゆえに、アニーリングを完了したステイプル(field)②の溶液をPEG沈殿、エタノール沈殿を行い生成する。
精製した結果field DNA origamiに悪影響が出るとも限らないので、その点も確認する。
②-1 PEG(ポリエチレングリコール)沈殿
水溶性のポリエーテルであるポリエチレングリコール(PEG)を用いたDNAの沈殿法であり、高分子量のDNAを優先的に沈殿させ(100bp以下)のDNAの除去が可能である。
<精製手順>
| PEG沈殿 溶液 |
|---|
| 50% ポリエチレングリコール 6μL |
| ステイプル(field)② 10μL |
| mQ 13.7μL |
| MgCl2(1M) 0.3μL |
| 計 30μL |
1.PEG沈殿溶液を遠心機に25°C 16.1rcfの条件で10分かける。
2.遠心後、上澄み液を取り除く。
3.10×TAE Mg2+を8μL加えてピペッティングを行う。
②-2 エタノール沈殿
エタノール(EtOH)は常温で水と任意の比率で混合できるが、DNAは溶解させない。ゆえにDNA溶液にエタノールを加えると、すでに溶けていたDNAはエタノールに溶けないため、次第に析出する。
しかしながら、DNAは負極性を持つため塩化ナトリウム溶液を加え、DNAを中和した後エタノールを加え、沈殿させる。
<精製手順>
| エタノール沈殿 溶液 |
|---|
| NaCl(5M) 10μL |
| ステイプル(field)② 90μL |
| 計 100μL |
1.サンプルに対し、0.1MになるようにNaClを加え、エタノール沈殿 溶液を作成する。15分ほど置く。
2.サンプル量の2倍の冷凍エタノールを加える。
3.4°C,15.000rpmの条件で20分遠心をかける。
4.上清を取り、70%エタノールを加え、やさしく混ぜる。
5.4°C,15.000rpmの条件で再度10分遠心にかける。
6.エタノールを乾かす。
7.同量以下のbuffer(1×TAE Mg2+)で戻す。
マーカー以外は6×Loading buffer 0.8μL Sample 4.2μL |
写真から見て分かるようにPEG沈
③引っ付けるためにどのような脂質を用いるか決める。
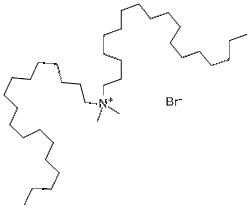
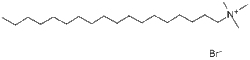
まず前提条件としてDNAはマイナスチャージを持つのでお互いは反発しあう。ゆえにこれらを引っ付けるためにはプラスチャージを持つ脂質が必要である。
そのため今回Dioctadecyldimethylammonium bromide (DODAB)もしくはStearyltrimethylammonium Bromide(SLLB)を用いる。
それぞれの化学式は以下。
 |
 |
④加える脂質のパラメータを決める。用いるDNAに対しての濃度の決定。
⑤脂質を加えて引っ付くかどうかを確認する。