Biotec 10. 박재형(JaeHyung Park): Difference between revisions
No edit summary |
No edit summary |
||
| (42 intermediate revisions by 2 users not shown) | |||
| Line 4: | Line 4: | ||
| • ∞ ♣ ♦ α β γ Δ ε η θ κ μ ν π ρ Σ τ φ ψ Ψ Ω ω<br> | | • ∞ ♣ ♦ α β γ Δ ε η θ κ μ ν π ρ Σ τ φ ψ Ψ Ω ω<br> | ||
가장 앞에 있는 기호가 이미지 파일에 사용되는 구분용 특수 문자 입니다. | 가장 앞에 있는 기호가 이미지 파일에 사용되는 구분용 특수 문자 입니다. | ||
=실험방법 회의= | |||
==7. 20== | |||
* '''Spin coating''' | |||
https://commons.wikimedia.org/wiki/File:SolGel_SpinCoating.jpg<br> | |||
[[Image:Pjh20150721-1.jpg]] | |||
#용액을 사용한 박막형성법 중 가장 널리 쓰이고 중요함 | |||
#회전하는 기판에 용액을 떨어뜨리면 기판의 각속도에 의해 용액의 대부분이 제거되고 박막이 잔류하게 됨 | |||
#두께, 형상, 표면조직의 재현성이 높으나 제한된 범위의 제어(두께)만 가능하며 다양한 변화를 주기는 어려움 | |||
#공정변수로는 기판의 회전속도, 용액의 점도 및 휘발성, 확산도, 고분자의 분자량, 농도 등이 있으며 용액의 양, 증착 속도, 시간 등은 영향을 적게 미침 | |||
#과량의 용액은 기판의 회전 동작으로 제거되므로 소량(0.1ml)의 용액만으로도 막을 얻을 수 있다. | |||
=실험방법 회의= | |||
==7. 20== | |||
http://link.springer.com/article/10.1007%2Fs12274-013-0291-0<br> | |||
[[Image:Pjh20150720-1.gif]] | |||
=Paper Study= | |||
==7. 16== | |||
===Chemiluminescence biosensors for DNA detection using graphene oxide and a horseradish peroxidase-mimicking DNAzyme=== | |||
http://www.researchgate.net/publication/51876132_Chemiluminescence_biosensors_for_DNA_detection_using_graphene_oxide_and_a_horseradish_peroxidase-mimicking_DNAzyme | |||
[http://www.researchgate.net/publication/51876132_Chemiluminescence_biosensors_for_DNA_detection_using_graphene_oxide_and_a_horseradish_peroxidase-mimicking_DNAzyme link title] | |||
* '''Chemiluminescence DNA detection''' | |||
[[Image:Pjh20150716-1.png]] | |||
#Horseradish peroxidase (HRP)-mimicking DNAzyme (HMDNAzyme)은 G-quadruplex structure에 hemin이 삽입되어 구성됨 | |||
#과산화수소와 luminol을 사용하여 Chemiluminescence(CL)을 일으킴 | |||
#HRP는 Protein peroxidase보다 DNA 서열이나 다른 타겟에 결합이 더 용이하며 그로인해 HMDNAzyme은 다양한 생체 분자의 비색 또는 CL 검출에 널리 적용 할 수있는 것을 의미 | |||
#Graphene oxide (GO)는 흑연의 얇은 시트 형태이며 형광 반응을 소광하는 기능을 가짐 | |||
#ssDNA와 GO는 서로 흡착되고 complementary target DNA 첨가 시 떨어짐 | |||
#따라서 CL의 강도는 ssDNA와 GO가 흡착되어 있을 때는 약하고 complementary target DNA가 첨가되어 ssDNA와 GO가 떨어졌을 때 강함 | |||
* '''Complementary target DNA(HT; target HIV sequence)와 GO 유무에 따른 관찰''' | |||
[[Image:Pjh20150716-2.png]] | |||
*(a)는 P-HIV(HMDNAzyme)만 존재하고 HT,GO 없음 | |||
*(b)는 P-HIV와 10 nM HT 존재 | |||
*(c)는 PHIV + 10 nM HT + 0.32 μg mL^-1 GO | |||
*(d)는 P-HIV + 0.32 μg mL^-1 GO | |||
*(e)는 buffer solution | |||
*Experimental conditions: P-HIV, 2 nM; luminol, 0.5 mM; H2O2, 30 mM | |||
#P-HIV가 단독으로 있거나 P-HIV와 HT만 존재할 때 가장 높은 CL 강도 나타냄 | |||
#P-HIV와 GO만 있을 때는 원래 CL 강도의 94%정도 감소 | |||
#P-HIV와 GO에 HT가 첨가 될 시에는 원래 CL강도의 46%정도까지 회복 | |||
*CL강도가 GO에 의해 소광되거나 GO의 흡착으로 인해 HMDNAzyme의 peroxidatic activity 떨어지는 것이 원인일 거라 추정 | |||
*전자는 확인할 수가 없어서 peroxidatic activity를 관찰하기 위해 비색법을 사용 | |||
[[Image:Pjh20150716-3.png]] | |||
*P-HIV는 0.125 μM로 동일 | |||
*(a)는 GO가 없음 | |||
*(b)는 0.005mg mL^-1 GO | |||
*(c)는 0.01mg mL^-1 GO | |||
*안쪽에 삽입된 그림은 ABTS를 사용한 비색법으로 흡광도를 관찰한 결과(λmax = 414 nm) | |||
[[Image:Pjh20150716-4.png]] | |||
#GO의 최소값인 0.32 μg mL^-1 으로 실험 | |||
#HT의 농도를 달리하여 실험(0, 0.1, 0.2, 0.8, 1.2, 1.5, 2, 2.5, 3nM) | |||
#optimal condition으로 실험하여 I = 646.609C + 14.7161의 방정식을 얻음(R^2 = 0.9914) | |||
[[Image:Pjh20150716-5.png]] | |||
#HT와 MT(single-base mismatched target), NT(non-complementary target)로 나눠 CL강도를 비교함 | |||
#HT에 비해 MT나 NT에서 CL강도가 낮음 | |||
#삽입 그림은 GO가 0.32 μg mL^-1 있을 때 HT,NT,MT를 비교 | |||
##(a)는 HT 존재, (b)는 MT 존재, (c)는 NT 존재, (d)는 HT,MT,NT 없음 | |||
* '''장점''' | |||
#HMDNAzyme는 fluorescent probe보다 제조하기 용이하며 비용이 싸다 | |||
#high sensitivity를 가지고 있어 34 pM target DNA도 검출 가능하여 더 복잡하고 정교한 실험을 하지 않아도 된다 | |||
#single-base mismatched target도 구별 가능하여 특정 타겟 서열을 구별할 수도 있다 | |||
=아이디어 회의= | |||
==7. 9== | |||
==A Sequence‐Selective Electrochemical DNA Biosensor Based on HRP‐Labeled Probe for Colorectal Cancer DNA Detection== | |||
http://www.tandfonline.com/doi/pdf/10.1080/00032710701746873#close <br> | |||
[http://www.tandfonline.com/doi/pdf/10.1080/00032710701746873 link title] | |||
* '''Crosslinking of Detection DNA with HRP''' | |||
- A reaction mixture was prepared by mixing 100 μl of detection DNA (72.5 μmol l^-1), 100 μl of glutaraldehyde (2.5%) and 200 μl of HRP (10 mg ml^-1, pH 7.00), and the reaction lasted for 7 h at 4℃. <br/> | |||
-Then 100 μl of L-Lysine (0.1%) was added to block for 3 h at 4℃. The reaction mixture was dialyzed overnight against 0.1 mol l^-1 phosphate buffer (pH 7.00) at 4℃ to remove the non-reacted low molecular reagents. | |||
=아이디어 회의= | |||
==7. 8== | |||
#생체 내 염 농도 0.9%, 약 0.154M | |||
#Seewater 논문 NaCl 농도 0.001,0.5,3M | |||
[[Image:2015-07-08 15.13.38.jpg|500px]] | |||
* '''(a)Biotin''' | |||
#Biotin은 avidin이라는 단백질에 높은 친화력을 가짐 | |||
#Biotin과의 반응에 의해 수정된 dUTP를 사용(T자리에 dUTP가 들어감) | |||
#Biotin으로 표지된 탐침의 위치는 형광물질이 결합된 아비딘으로 차리하여 검정 | |||
* '''(b)Horseradish peroxidase(HRP)''' | |||
#Horseradish peroxidase and luminol | |||
Horseradish pero×idase(HRP)는 luminol 을 분해하여 빛을 내게 함(chemiluminescence) | |||
#Glutaraldehyde 를 이용해 HRP를 ssDNA probe 에 결합시킨 뒤 이를 ssDNA에 처리하고 luminol을 넣어주면 혼성화 된 자리에서 luminol이 분해되며 발광함. 이는 일반사진 필름을 이용해서도 탐지할 수 있음 | |||
* ''Luminol'' | |||
#화학식 C8H7N3O2, 분자량 177.16, 녹는점 319~320℃ | |||
#산화하면 화학발광, 즉 형광을 발생하는 특징이 있는 백색 결정 | |||
#이것을 사용하여 구리, 철 등의 검출이나 과산화수소(활성산소),시안이온을 정량할 수 있다 | |||
#또한 혈액(헤모글로빈이나 헤민)에 의해 예민하게 발광하기 때문에 혈흔 감식에 사용되고 있다. | |||
[[Image:Pjh20150709-1.png]] | |||
#루미놀이 수산화 이온과 반응하면, 2가 음이온이 형성 | |||
#과산화수소에서 산소가 발생하고 루미놀 이온과 반응 | |||
#이 반응의 생성물인 유기 과산화물(organic peroxide)은 질소가 빠짐으로써 만들어지는데 매우 불안정한 상태 | |||
#전자가 이 때 들뜬 상태에 있다가 바닥 상태로 전이되며 에너지를 방출 | |||
#이 에너지의 방출이 파란색으로 보인다. | |||
=Paper Study= | |||
==7. 6== | |||
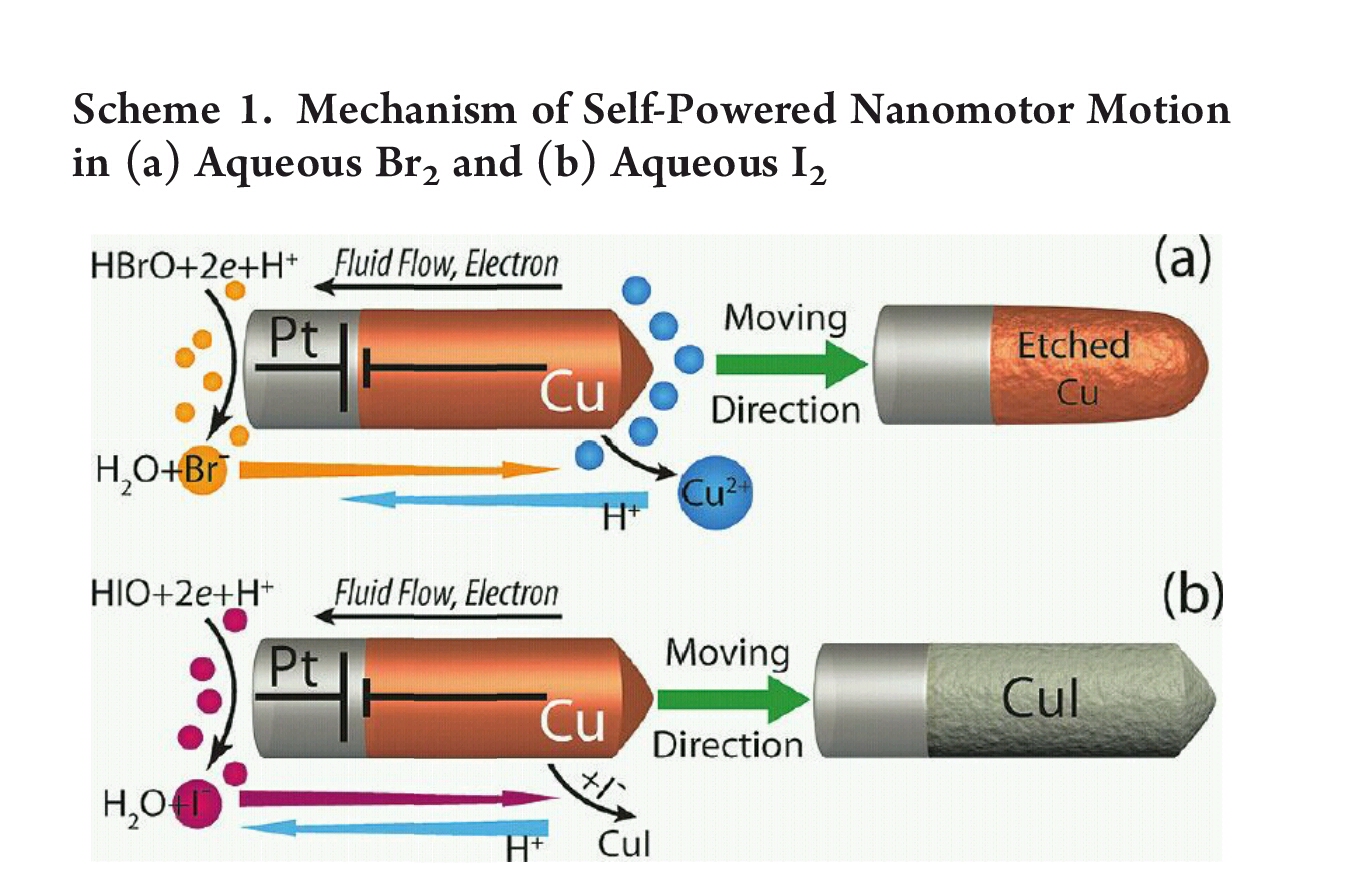
===Autonomous Nanomotor Based on CopperÀPlatinum Segmented Nanobattery=== | |||
[http://pubs.acs.org/doi/abs/10.1021/ja2082735 link title] | |||
[[Image:2015-07-06 12.21.25.jpg]] | |||
#버블이 나오지 않아서 관찰이 용이함 | |||
#TEM으로 Cu와 Pt 구분 가능 | |||
#구리의 길이로 속도 조절 가능 | |||
=아이디어 회의= | |||
==7. 4== | |||
#본체가 나노미터일 경우 닻을 DNA로 하면 크기의 차이가 너무 큼 | |||
#유기용매에서는 DNA가 용해되지 않음 | |||
#동력원: Pt,Mg, 전극을 이용한 nanobattery | |||
==7. 3== | |||
예비군 훈련 | |||
=아이디어 관련 공부= | |||
==7. 2== | |||
* '''Terminal tranferase''' | |||
[[Image:7071 1.jpg]] | |||
#DNA homopolymer의 꼬리를 신장시키는 기능을 가지고 있음 | |||
#3'-OH에 deoxynuleotide triphosphate를 붙게하여 신장시킴 | |||
#EcoRI과 같은 제한효소로 인해 생성된 sticky end를 신장시키는 것도 가능 | |||
#약 10~40개의 nucleotide를 추가함 | |||
=아이디어 회의= | |||
==7. 1== | |||
#Si02와 Au를 이용한 모터 | |||
##linker에 DNA sheet를 부착하여 돛의 기능을 할 수 있을까? | |||
#track을 휘어지게 하거나 트랙을 나누어서 위아래로 트랙이 있을 경우 motor의 키가 커지게 하여 위쪽트랙과 아래쪽 트랙으로 모터가 이동할 수 있을까? | |||
[[Image:Pjh20150701.png]] | |||
=아이디어 회의= | |||
==6. 30== | |||
#DNA 17mer을 이용하여 세로구조로 키(높이)가 변하며 이동하는 모터 | |||
#네가닥의 발로 회전하며 이동하는 모터 | |||
=Paper Study= | =Paper Study= | ||
==6. 29== | ==6. 29== | ||
===A Synthetic DNA Walker for Molecular Transport=== | ===A Synthetic DNA Walker for Molecular Transport=== | ||
http://pubs.acs.org/doi/abs/10.1021/ja047543j#close <br> | |||
[http://pubs.acs.org/doi/abs/10.1021/ja047543j link title] | |||
[[Image:Pjh20150629-1.png]] | |||
* '''DNA Sequences for the Walker System''' | |||
#walker와 track strands, attachment strands 서열들 | |||
#walker에는 소광제 존재하며 track에는 형광제가 있음 | |||
[[Image:Pjh20150629-2.png]] | |||
* '''Schematic of walker locomotion''' | |||
#walker의 1번 다리(W1)가 attachment strand(A1)에 의해 track strand(T1)에 결합 | |||
#마찬가지로 A2로 인해 W2가 T2와 결합 | |||
#detachment fuel strands(D1)에 의해 D1과 A1이 결합함으로써 W1과 T1사이의 결합이 풀림 | |||
#위와 같은 cycle로 반복됨 | |||
[[Image:Pjh20150629-3.png]] | |||
* '''Non-denaturing polyacrylamide gel analysis & Multiplexed real-time fluorescence monitoring''' | |||
#전기영동의 밴드를 통해 결합 유무 확인 | |||
#Multiplexed real-time fluorescence monitoring을 통해 각 단계의 형광 반응을 확인 | |||
#예를 들어 W1과 T1의 결합 시 소광제로 인해 T1의 형광 반응이 감소 -> D1의 첨가로 인해 결합이 해제되면 다시 T1의 형광반응 증가 | |||
=Paper Study= | =Paper Study= | ||
==6. 25== | ==6. 25== | ||
===A Precisely Controlled DNA Biped Walking Device=== | ===A Precisely Controlled DNA Biped Walking Device=== | ||
http://pubs.acs.org/doi/abs/10.1021/nl049527q#close <br> | |||
[http://pubs.acs.org/doi/abs/10.1021/nl049527q link title] | |||
[[Image:Pjh20150625-1.png]] | |||
* '''Cartoon of the biped system with DNA base sequence included''' | |||
#DNA로 이루어진 DNA Biped Walking Device와 Track | |||
#DNA Biped Walking Device는 느슨한 Linker(DNA)로 연결됨 | |||
#Foot 부분(F1,F2)과 트랙의 Foothold 부분은 single strand로 이루어짐 | |||
#Foot 부분(F1,F2)의 끝에는 Psoralen groups이 존재 | |||
#Foot과 Foothold를 연결하기 위해 set strands(SS1A,SS2B)를 사용 | |||
[[Image:Pjh20150625-2.png]] | |||
* '''Cartoon depiction of biped system taking a full step''' | |||
#Foot과 Foothold를 연결하는 set strand를 떼어내기 위해 unset strand를 사용 | |||
#set strand 떨어져 나가면 발은 자유로워짐 | |||
#다음 foothold와 연결하기 위해 set strand를 넣어주면 다음 foothold와 foot이 결합하여 전진 | |||
#뒤쪽의 foot도 마찬가지의 시스템으로 이동 | |||
[[Image:Pjh20150625-3.png]] | |||
* '''Shows a detailed picture of a foot strand (tan), a set strand (black), and a foothold strand (blue)''' | |||
#psoralen에 UV를 조사할 시 set strand,foothold strand에 연결이 이루어 질 수 있음 | |||
#set strand,foothold strand에 있는 T에 psoralen이 결합 가능 | |||
#foot strand,set strand,foothold strand가 모두 결합한 형태가 (b), 실패하고 두가지만 붙은 것이 (c)와 (d) | |||
[[Image:Pjh20150625-4.png]] | |||
* '''Record of a biped walk''' | |||
#psoralen의 결합(3 component complexes)를 통한 walking의 확인 | |||
#set strand가 부착 시 foot 끝 부분의 psoralen에 UV를 조사하면 3 component complexes가 일어나게 됨 | |||
#3 component complexes의 형성을 관찰하여 움직임을 확인 | |||
=Paper Study= | =Paper Study= | ||
==6. 24== | ==6. 24== | ||
===A Single DNA Molecule Nanomotor=== | ===A Single DNA Molecule Nanomotor=== | ||
http://pubs.acs.org/doi/abs/10.1021/nl015713+#close <br> | |||
[http://pubs.acs.org/doi/abs/10.1021/nl015713+ link title] | |||
[[Image:Pjh20150624-1.png]] | |||
* '''Structure and operation of a Single DNA Molecule Nanomotor''' | |||
#Motor는 17mer DNA nanomotor (T1G2G3T4T5G6G7T8G9T10G11G12T13T14G15G16T17)로 구성됨 | |||
#17mer DNA nanomotor는 tetraplex (TE) 형태로 바뀔 수 있음 | |||
#17mer DNA nanomotor의 양쪽 끝부분에는 형광제와 소광제가 있음 | |||
#17mer DNA와 상보적인 strand α가 주입될 시 17mer DNA가 팽창하여 duplex (DU)형태로 바뀜 | |||
#strand α는 strand β가 주입되면 strand α와 결합하여 Motor에서 떨어져 나감, 다시 원래 형태(TE)로 바뀜 | |||
#이러한 시스템이 순환 | |||
[[Image:Pjh20150624-2.png]] | |||
* '''Fluorescent response of the nanomotor 17mer (red) and the S17mer (green)''' | |||
#S17mer는 nanomotor 17mer와 유사한 비교군 | |||
#K+는 G-rich sequence를 loose random coil로부터 compact TE 형태로 변환 | |||
#(I)는 기본상태 (II)는 K+첨가 (III)는 complementary sequences 첨가 | |||
#K+를 첨가하면 compact TE로 변환되기 때문에 형광반응이 감소 | |||
#complementary sequences 첨가 시 DU상태가 되므로 형광반응 증가 | |||
[[Image:Pjh20150624-3.png]] | |||
* '''Cycling of the DNA nanomotor''' | |||
#nanomotor가 순환됨에 따른 형광반응을 측정 | |||
| Line 27: | Line 272: | ||
==6. 23== | ==6. 23== | ||
===An Autonomous DNA Nanomotor Powered by a DNA Enzyme=== | ===An Autonomous DNA Nanomotor Powered by a DNA Enzyme=== | ||
http://onlinelibrary.wiley.com/doi/10.1002/anie.200453779/abstract#close <br> | |||
[http://onlinelibrary.wiley.com/doi/10.1002/anie.200453779/abstract link title] | |||
[[Image:Pjh20150623-1.png]] | |||
* '''Structure and operation of an autonomous DNA nanomotor''' | |||
#E,F 두 가닥으로 구성된 모터 | |||
#E strand에는 RNA-cleaving10-23 DNA enzyme가 있음 | |||
#F strand는 두 가지의 fluorophores가 존재함 | |||
#The enzyme substrate(S)는 DNA–RNA chimera임 | |||
#E strand가 수축상태(close)에서 S가 결합함으로써 팽창상태(open)가 됨 | |||
#S는 RNA-cleaving10-23 DNA enzyme으로 인해 절단됨 | |||
#S는 절단되어 S1,S2로 나뉘어졌다가 E strand에서 떨어져 나감 | |||
#이러한 원리로 open/close 시스템이 순환 | |||
#이 때 F strand의 fluorophores가 반응함 | |||
[[Image:Pjh20150623-2.png]] | |||
* '''Native gel electrophoretic analysis of DNA motor formation''' | |||
#전기영동으로 Motor의 형태 확인 | |||
#S`는 S와 유사하지만 절단되지 않음 | |||
#S가 Motor와 결합한 뒤 S1,S2로 절단됨을 확인 | |||
[[Image:Pjh20150623-3.png]] | |||
* '''The autonomous cycling of the DNA motor & Fluorescence spectra of the DNA motor in the closed and open states''' | |||
#Motor과 S는 1:20의 비율로 첨가 | |||
#20개의 S가 30분에 모두 분해됨, 1개당 1.5분의 시간 걸림 | |||
#Open/Close의 순환 시스템을 형광반응을 통해 확인 | |||
Latest revision as of 22:08, 20 July 2015
- 특수문자들
| • ∞ ♣ ♦ α β γ Δ ε η θ κ μ ν π ρ Σ τ φ ψ Ψ Ω ω
가장 앞에 있는 기호가 이미지 파일에 사용되는 구분용 특수 문자 입니다.
실험방법 회의
7. 20
- Spin coating
https://commons.wikimedia.org/wiki/File:SolGel_SpinCoating.jpg

- 용액을 사용한 박막형성법 중 가장 널리 쓰이고 중요함
- 회전하는 기판에 용액을 떨어뜨리면 기판의 각속도에 의해 용액의 대부분이 제거되고 박막이 잔류하게 됨
- 두께, 형상, 표면조직의 재현성이 높으나 제한된 범위의 제어(두께)만 가능하며 다양한 변화를 주기는 어려움
- 공정변수로는 기판의 회전속도, 용액의 점도 및 휘발성, 확산도, 고분자의 분자량, 농도 등이 있으며 용액의 양, 증착 속도, 시간 등은 영향을 적게 미침
- 과량의 용액은 기판의 회전 동작으로 제거되므로 소량(0.1ml)의 용액만으로도 막을 얻을 수 있다.
실험방법 회의
7. 20
http://link.springer.com/article/10.1007%2Fs12274-013-0291-0

Paper Study
7. 16
Chemiluminescence biosensors for DNA detection using graphene oxide and a horseradish peroxidase-mimicking DNAzyme
http://www.researchgate.net/publication/51876132_Chemiluminescence_biosensors_for_DNA_detection_using_graphene_oxide_and_a_horseradish_peroxidase-mimicking_DNAzyme link title
- Chemiluminescence DNA detection
- Horseradish peroxidase (HRP)-mimicking DNAzyme (HMDNAzyme)은 G-quadruplex structure에 hemin이 삽입되어 구성됨
- 과산화수소와 luminol을 사용하여 Chemiluminescence(CL)을 일으킴
- HRP는 Protein peroxidase보다 DNA 서열이나 다른 타겟에 결합이 더 용이하며 그로인해 HMDNAzyme은 다양한 생체 분자의 비색 또는 CL 검출에 널리 적용 할 수있는 것을 의미
- Graphene oxide (GO)는 흑연의 얇은 시트 형태이며 형광 반응을 소광하는 기능을 가짐
- ssDNA와 GO는 서로 흡착되고 complementary target DNA 첨가 시 떨어짐
- 따라서 CL의 강도는 ssDNA와 GO가 흡착되어 있을 때는 약하고 complementary target DNA가 첨가되어 ssDNA와 GO가 떨어졌을 때 강함
- Complementary target DNA(HT; target HIV sequence)와 GO 유무에 따른 관찰
- (a)는 P-HIV(HMDNAzyme)만 존재하고 HT,GO 없음
- (b)는 P-HIV와 10 nM HT 존재
- (c)는 PHIV + 10 nM HT + 0.32 μg mL^-1 GO
- (d)는 P-HIV + 0.32 μg mL^-1 GO
- (e)는 buffer solution
- Experimental conditions: P-HIV, 2 nM; luminol, 0.5 mM; H2O2, 30 mM
- P-HIV가 단독으로 있거나 P-HIV와 HT만 존재할 때 가장 높은 CL 강도 나타냄
- P-HIV와 GO만 있을 때는 원래 CL 강도의 94%정도 감소
- P-HIV와 GO에 HT가 첨가 될 시에는 원래 CL강도의 46%정도까지 회복
- CL강도가 GO에 의해 소광되거나 GO의 흡착으로 인해 HMDNAzyme의 peroxidatic activity 떨어지는 것이 원인일 거라 추정
- 전자는 확인할 수가 없어서 peroxidatic activity를 관찰하기 위해 비색법을 사용
- P-HIV는 0.125 μM로 동일
- (a)는 GO가 없음
- (b)는 0.005mg mL^-1 GO
- (c)는 0.01mg mL^-1 GO
- 안쪽에 삽입된 그림은 ABTS를 사용한 비색법으로 흡광도를 관찰한 결과(λmax = 414 nm)
- GO의 최소값인 0.32 μg mL^-1 으로 실험
- HT의 농도를 달리하여 실험(0, 0.1, 0.2, 0.8, 1.2, 1.5, 2, 2.5, 3nM)
- optimal condition으로 실험하여 I = 646.609C + 14.7161의 방정식을 얻음(R^2 = 0.9914)
- HT와 MT(single-base mismatched target), NT(non-complementary target)로 나눠 CL강도를 비교함
- HT에 비해 MT나 NT에서 CL강도가 낮음
- 삽입 그림은 GO가 0.32 μg mL^-1 있을 때 HT,NT,MT를 비교
- (a)는 HT 존재, (b)는 MT 존재, (c)는 NT 존재, (d)는 HT,MT,NT 없음
- 장점
- HMDNAzyme는 fluorescent probe보다 제조하기 용이하며 비용이 싸다
- high sensitivity를 가지고 있어 34 pM target DNA도 검출 가능하여 더 복잡하고 정교한 실험을 하지 않아도 된다
- single-base mismatched target도 구별 가능하여 특정 타겟 서열을 구별할 수도 있다
아이디어 회의
7. 9
A Sequence‐Selective Electrochemical DNA Biosensor Based on HRP‐Labeled Probe for Colorectal Cancer DNA Detection
http://www.tandfonline.com/doi/pdf/10.1080/00032710701746873#close
link title
- Crosslinking of Detection DNA with HRP
- A reaction mixture was prepared by mixing 100 μl of detection DNA (72.5 μmol l^-1), 100 μl of glutaraldehyde (2.5%) and 200 μl of HRP (10 mg ml^-1, pH 7.00), and the reaction lasted for 7 h at 4℃.
-Then 100 μl of L-Lysine (0.1%) was added to block for 3 h at 4℃. The reaction mixture was dialyzed overnight against 0.1 mol l^-1 phosphate buffer (pH 7.00) at 4℃ to remove the non-reacted low molecular reagents.
아이디어 회의
7. 8
- 생체 내 염 농도 0.9%, 약 0.154M
- Seewater 논문 NaCl 농도 0.001,0.5,3M
- (a)Biotin
- Biotin은 avidin이라는 단백질에 높은 친화력을 가짐
- Biotin과의 반응에 의해 수정된 dUTP를 사용(T자리에 dUTP가 들어감)
- Biotin으로 표지된 탐침의 위치는 형광물질이 결합된 아비딘으로 차리하여 검정
- (b)Horseradish peroxidase(HRP)
- Horseradish peroxidase and luminol
Horseradish pero×idase(HRP)는 luminol 을 분해하여 빛을 내게 함(chemiluminescence)
- Glutaraldehyde 를 이용해 HRP를 ssDNA probe 에 결합시킨 뒤 이를 ssDNA에 처리하고 luminol을 넣어주면 혼성화 된 자리에서 luminol이 분해되며 발광함. 이는 일반사진 필름을 이용해서도 탐지할 수 있음
- Luminol
- 화학식 C8H7N3O2, 분자량 177.16, 녹는점 319~320℃
- 산화하면 화학발광, 즉 형광을 발생하는 특징이 있는 백색 결정
- 이것을 사용하여 구리, 철 등의 검출이나 과산화수소(활성산소),시안이온을 정량할 수 있다
- 또한 혈액(헤모글로빈이나 헤민)에 의해 예민하게 발광하기 때문에 혈흔 감식에 사용되고 있다.
- 루미놀이 수산화 이온과 반응하면, 2가 음이온이 형성
- 과산화수소에서 산소가 발생하고 루미놀 이온과 반응
- 이 반응의 생성물인 유기 과산화물(organic peroxide)은 질소가 빠짐으로써 만들어지는데 매우 불안정한 상태
- 전자가 이 때 들뜬 상태에 있다가 바닥 상태로 전이되며 에너지를 방출
- 이 에너지의 방출이 파란색으로 보인다.
Paper Study
7. 6
Autonomous Nanomotor Based on CopperÀPlatinum Segmented Nanobattery
- 버블이 나오지 않아서 관찰이 용이함
- TEM으로 Cu와 Pt 구분 가능
- 구리의 길이로 속도 조절 가능
아이디어 회의
7. 4
- 본체가 나노미터일 경우 닻을 DNA로 하면 크기의 차이가 너무 큼
- 유기용매에서는 DNA가 용해되지 않음
- 동력원: Pt,Mg, 전극을 이용한 nanobattery
7. 3
예비군 훈련
아이디어 관련 공부
7. 2
- Terminal tranferase
- DNA homopolymer의 꼬리를 신장시키는 기능을 가지고 있음
- 3'-OH에 deoxynuleotide triphosphate를 붙게하여 신장시킴
- EcoRI과 같은 제한효소로 인해 생성된 sticky end를 신장시키는 것도 가능
- 약 10~40개의 nucleotide를 추가함
아이디어 회의
7. 1
- Si02와 Au를 이용한 모터
- linker에 DNA sheet를 부착하여 돛의 기능을 할 수 있을까?
- track을 휘어지게 하거나 트랙을 나누어서 위아래로 트랙이 있을 경우 motor의 키가 커지게 하여 위쪽트랙과 아래쪽 트랙으로 모터가 이동할 수 있을까?
아이디어 회의
6. 30
- DNA 17mer을 이용하여 세로구조로 키(높이)가 변하며 이동하는 모터
- 네가닥의 발로 회전하며 이동하는 모터
Paper Study
6. 29
A Synthetic DNA Walker for Molecular Transport
http://pubs.acs.org/doi/abs/10.1021/ja047543j#close
link title
- DNA Sequences for the Walker System
- walker와 track strands, attachment strands 서열들
- walker에는 소광제 존재하며 track에는 형광제가 있음
- Schematic of walker locomotion
- walker의 1번 다리(W1)가 attachment strand(A1)에 의해 track strand(T1)에 결합
- 마찬가지로 A2로 인해 W2가 T2와 결합
- detachment fuel strands(D1)에 의해 D1과 A1이 결합함으로써 W1과 T1사이의 결합이 풀림
- 위와 같은 cycle로 반복됨
- Non-denaturing polyacrylamide gel analysis & Multiplexed real-time fluorescence monitoring
- 전기영동의 밴드를 통해 결합 유무 확인
- Multiplexed real-time fluorescence monitoring을 통해 각 단계의 형광 반응을 확인
- 예를 들어 W1과 T1의 결합 시 소광제로 인해 T1의 형광 반응이 감소 -> D1의 첨가로 인해 결합이 해제되면 다시 T1의 형광반응 증가
Paper Study
6. 25
A Precisely Controlled DNA Biped Walking Device
http://pubs.acs.org/doi/abs/10.1021/nl049527q#close
link title
- Cartoon of the biped system with DNA base sequence included
- DNA로 이루어진 DNA Biped Walking Device와 Track
- DNA Biped Walking Device는 느슨한 Linker(DNA)로 연결됨
- Foot 부분(F1,F2)과 트랙의 Foothold 부분은 single strand로 이루어짐
- Foot 부분(F1,F2)의 끝에는 Psoralen groups이 존재
- Foot과 Foothold를 연결하기 위해 set strands(SS1A,SS2B)를 사용
- Cartoon depiction of biped system taking a full step
- Foot과 Foothold를 연결하는 set strand를 떼어내기 위해 unset strand를 사용
- set strand 떨어져 나가면 발은 자유로워짐
- 다음 foothold와 연결하기 위해 set strand를 넣어주면 다음 foothold와 foot이 결합하여 전진
- 뒤쪽의 foot도 마찬가지의 시스템으로 이동
- Shows a detailed picture of a foot strand (tan), a set strand (black), and a foothold strand (blue)
- psoralen에 UV를 조사할 시 set strand,foothold strand에 연결이 이루어 질 수 있음
- set strand,foothold strand에 있는 T에 psoralen이 결합 가능
- foot strand,set strand,foothold strand가 모두 결합한 형태가 (b), 실패하고 두가지만 붙은 것이 (c)와 (d)
- Record of a biped walk
- psoralen의 결합(3 component complexes)를 통한 walking의 확인
- set strand가 부착 시 foot 끝 부분의 psoralen에 UV를 조사하면 3 component complexes가 일어나게 됨
- 3 component complexes의 형성을 관찰하여 움직임을 확인
Paper Study
6. 24
A Single DNA Molecule Nanomotor
http://pubs.acs.org/doi/abs/10.1021/nl015713+#close
link title
- Structure and operation of a Single DNA Molecule Nanomotor
- Motor는 17mer DNA nanomotor (T1G2G3T4T5G6G7T8G9T10G11G12T13T14G15G16T17)로 구성됨
- 17mer DNA nanomotor는 tetraplex (TE) 형태로 바뀔 수 있음
- 17mer DNA nanomotor의 양쪽 끝부분에는 형광제와 소광제가 있음
- 17mer DNA와 상보적인 strand α가 주입될 시 17mer DNA가 팽창하여 duplex (DU)형태로 바뀜
- strand α는 strand β가 주입되면 strand α와 결합하여 Motor에서 떨어져 나감, 다시 원래 형태(TE)로 바뀜
- 이러한 시스템이 순환
- Fluorescent response of the nanomotor 17mer (red) and the S17mer (green)
- S17mer는 nanomotor 17mer와 유사한 비교군
- K+는 G-rich sequence를 loose random coil로부터 compact TE 형태로 변환
- (I)는 기본상태 (II)는 K+첨가 (III)는 complementary sequences 첨가
- K+를 첨가하면 compact TE로 변환되기 때문에 형광반응이 감소
- complementary sequences 첨가 시 DU상태가 되므로 형광반응 증가
- Cycling of the DNA nanomotor
- nanomotor가 순환됨에 따른 형광반응을 측정
Paper Study
6. 23
An Autonomous DNA Nanomotor Powered by a DNA Enzyme
http://onlinelibrary.wiley.com/doi/10.1002/anie.200453779/abstract#close
link title
- Structure and operation of an autonomous DNA nanomotor
- E,F 두 가닥으로 구성된 모터
- E strand에는 RNA-cleaving10-23 DNA enzyme가 있음
- F strand는 두 가지의 fluorophores가 존재함
- The enzyme substrate(S)는 DNA–RNA chimera임
- E strand가 수축상태(close)에서 S가 결합함으로써 팽창상태(open)가 됨
- S는 RNA-cleaving10-23 DNA enzyme으로 인해 절단됨
- S는 절단되어 S1,S2로 나뉘어졌다가 E strand에서 떨어져 나감
- 이러한 원리로 open/close 시스템이 순환
- 이 때 F strand의 fluorophores가 반응함
- Native gel electrophoretic analysis of DNA motor formation
- 전기영동으로 Motor의 형태 확인
- S`는 S와 유사하지만 절단되지 않음
- S가 Motor와 결합한 뒤 S1,S2로 절단됨을 확인
- The autonomous cycling of the DNA motor & Fluorescence spectra of the DNA motor in the closed and open states
- Motor과 S는 1:20의 비율로 첨가
- 20개의 S가 30분에 모두 분해됨, 1개당 1.5분의 시간 걸림
- Open/Close의 순환 시스템을 형광반응을 통해 확인
Paper Study
2. 24 (Tue)
Self-assembled DNA nanostructures for distance-dependent multivalent ligand–protein binding
http://www.nature.com/nnano/journal/v3/n7/abs/nnano.2008.164.html#close
link title
- Aptamer
- Aptamers are oligonucleotide or peptide molecules that bind to a specific target molecule.
- Aptamer의 거리,helix 수에 따른 결합 효율
- aptamer 거리가 5.3nm일 때 최적
- four-helix bundle(4HB)가 5HB보다 결합이 높음
- Aptamer의 종류에 따른 효율
- 서로다른 Aptamer일 때가 동일한 aptamer일 때보다 효율이 높음
- Aptamer의 거리를 통한 실험결과
- Aptamer의 거리가 5.8nm,20.7nm 인 두가지로 실험을 진행했을 때 5.8nm인 부분에 Thrombin이 결합
- ※즉 선택적인 결합이 가능
Paper Study
2. 17 (Tue)
Single-molecule chemical reactions on DNA origami
http://www.nature.com/nnano/journal/v5/n3/full/nnano.2010.5.html#close
link title
- Linker A,B,C
- non-cleavable linker type A
- linker type B, which contains a disulphide moiety that can be cleaved by reduction
- Linker C can be cleaved by singlet oxygen generated with light in the presence of a singlet oxygen photosensitizer (PS)
- three functional groups
- The reaction of biotin-linked azide Az took place in the presence of the in situ generated copper(I)-THTA (tris-(1-[3-hydroxypropyl]triazolyl-4-methyl)amine) catalyst
- The reaction of the biotin NHS-ester Es was performed in a slightly alkaline buffer/DMF mixture
- The reaction of the biotin-linked alkyne Al was studied in the presence of the in situ generated copper(I)-THTA in a DMF/buffer mixture
- The three reactions can proceed successively on the immobilized DNA origami template with high selectivity.
Team presentation
2. 12 (Thu)
Nucleic acid enzymes
Box file name : 150212_Team3-nucleic acid enzymes.pptx
- PCR
- DNA,RNA의 특정영역을 대량으로 증폭하는 기술.
- -Primer가 taq polymerase에 의해 신장됨.
- Denaturation - Annealing - Elongation 의 과정을 반복하여 수행.
- -이론적으로 n사이클 시행 시 2^n개의 ds DNA 생성.
- mRNA를 cDNA로 역전사하는 reverse transcriptase를 이용하는 RT-PCR도 있음.
- Restriction Enzyme
- ds DNA의 특정 염기서열을 인식하여 그 부분이나 그 주변을 절단하는 효소.
- 바이러스 등 외부에서 온 DNA를 절단하여 배제시키는 자기방어 기능도 가짐.
- Restriction Enzyme에 의해 절단되는 모양에 따라 Blunt end,Sticky end로 나뉨
- Ligation
- DNA나 RNA의 끝을 이어주는 과정
- Restriction Enzyme과 함께 쓰이는 경우가 많음.
- Ligase에 의해 phosphodiester bond로 DNA 분자 내에서 일어나는 한가닥 파손(불연속)을 복구.
- Gene Cloning
- PCR을 통해 foreign DNA을 생성하고
- 같은 Restriction Enzyme으로 vector와 foreign DNA을 절단한 뒤
- foreign DNA을 vector에 삽입하여 Ligation시켜 결합함.
- 만들어진 recombinant DNA를 bacteria 내부로 trasformation 시키면
- cell 내에서 증식이 일어나고 셀 자체도 많은 colony를 이루게 됨.
Paper Study
2. 10 (Tue)
Molecularly self-assembled nucleic acid nanoparticles for targeted in vivo siRNA delivery
http://www.nature.com/nnano/journal/v7/n6/abs/nnano.2012.73.html#close
link title
- siRNA(small interfering RNAs)
- 이중가닥의 RNA(double-stranded RNA)가 Dicer에 의해 절단되어 생성되는 21에서 25nt 크기의 작은 RNA조각
- 상보적인 서열을 갖는 mRNA에 특이적으로 결합하여 단백질 발현을 억제
- siRNA 가 mRNA와 100% 상보적인 경우는 mRNA 를 분해하고, 약 80-90% 상보적인 경우는 번역을 억제
- self-assembly of the DNA tetrahedron
- cell 내로 siRNA 전달 가능
- 크기를 정의할 수 있어야한다.
- 높이는 8nm, 모서리 길이는 10nm
- 리간드가 적절한 공간배향에 있을 때 gene silencing이 이뤄진다.
- 전달 효율
- siRNA의 circulation time (t1/2 ≈ 6 min), ONPs의 circulation time (t1/2 ≈ 24.2 min)
- ONPs가 only siRNA일 때보다 세포 내로 siRNA의 전달이 더 원활하게 이뤄진다.
Team presentation
2. 5 (Thu)
Gel electrophoresis
Box file name : Team3_150205_gel-eletrophoresis.pptx
- Gel electrophoresis
- (-)charge를 띤 입자가 gel을 통해 (+)방향으로 이동
- 이동속도는 입자의 전하량,크기,구조에 따라 달라짐.
- Agarose gel electrophoresis
- DNA,RNA를 electrophoresis 할 때 주로 사용
- 핵산이 가지고 있는 전하를 이용하여 agarose gel상에서 크기,구조에 따라 분리
- electrophoresis 후 EtBr 처리하고 UV를 통해 Band 관찰
- SDS-PAGE
- Protein을 electrophoresis 할 때 주로 사용
- Protein은 전하, 구조가 일정치 않으므로 sds와 dtt를 처리하여 변성시킴
- Polyacrylamide gel은 disc gel로 stacking gel과 running gel로 이루어짐
- Gly와 Cl에 의해 protein이 stacking gel에서 일직선상에 정렬되고 running gel에서 size에 의한 분리가 일어남
- electrophoresis 후 Coomassie Brilliant Blue로 염색하고 Band 관찰
- Marker를 통해 size의 비교. -(agarose gel electrophoresis도 동일)
- 분리능을 높이기 위해 Two-dimentinal electrophoresis 또는 Western blot 하기도 함
caDNAno
2. 1 (Sun)
Paper Study
2. 1 (Sun)
Precision Templating with DNA of a Virus-like Particle with Peptide Nanostructures
http://pubs.acs.org/doi/abs/10.1021/ja4008003#close Box file name : Ruff2013J_Am_Chem_Soc(jae hyung park).pdf
link title
- 정확한 길이의 빌딩블록을 자가조립하는 것은 쉽지않다.
- 섬유형 바이러스의 모양을 모방하여 정밀하게 길이를 제어할 수 있다.
- 인공 capsomer를 만든다.
- 인공 capsomer의 양전하 부분에 주형 DNA가 결합하여 섬유형 바이러스 같은 형태를 만든다.
- 주형 DNA에 의해서 길이를 정밀하게 control할 수 있다.
- 정확한 길이의 초분자의 활용
- 특정 크기의 물질을 적재하거나 더 복잡한 구조의 template, building block을 만들 수 있다.
- 정확한 크기의 초분자를 만들 수 있음으로서 새로운 물질을 디자인하는데 쓰일 수 있다.
caDNAno
1. 22 (Thu)
Paper Study
1. 20 (Tue)
End-joining long nucleic acid polymers - STV
http://nar.oxfordjournals.org/content/36/16/e104.short#close Box file name : Van_den_Hout2008Nucleic_Acids_Res(Jaehyung Park).pdf, PJH150120_stv.pptx
link title
- Ligation
DNA나 RNA의 끝을 이어주는 과정 single-strand, long-nucleic acid일 경우 효율이 떨어지는 단점
- Biotin–streptavidin linkage
streptavidin은 단백질의 일종 biotin은 비타민의 일종으로
강한 결합력으로 결합함.
강한 결합력을 이용해 end-joining을 하는 방법.
2-step으로 이루어짐 한분자에 stv를 붙이고(1-step)
unbound stv를 제거한 다음 두번째 분자를 붙임(2-step)
- Gel electrophoresis
전극을 이용하여 물질을 이동시키며
크기나 구조에 따라 이동속도의 차이가 일어남.
크기를 분석하는 방법.
- The application of Biotin–streptavidin linkage
single-strand를 필요로 하는 실험에 쓰임.
강한 결합력을 이용하여 병원성 박테리아의 선택적 포획 등에도 쓰임.
Paper Study
1. 3 (Sat)
Folding DNA to create nanoscale shapes and patterns - DNA origami
http://www.nature.com/nature/journal/v440/n7082/full/nature04586.html#close Box file name : Nature04586(PJH).pdf, PJH150103_dna origami.pptx
link title
- DNA Structure
- DNA is double helix structure.
- In DNA, there are four different types of nitrogenous base.
According to base pairing rules (A with T and C with G), hydrogen bonds bind the nitrogenous bases of the two separate polynucleotide strands to make double-stranded DNA.- A is for adenine
- G is for guanine
- C is for cytosine
- T is for thymine
- DNA origami
- DNA origami is the nanoscale folding of DNA to create arbitrary two- and three-dimensional shapes at the nanoscale.
- A long single strand DNA and staple DNA strand are required
- The use of a long single strand DNA in M13mp18.
- Staple DNA strand folded long single strand DNA.
- Use a computer to determine the way to create the correct staples needed to form a certain shape. And we create arbitrary two- and three-dimensional shapes at the nanoscale.
- The application of DNA origami
- DNA origami will enable making small computer.
- DNA origami be used to create nanorobots capable of finding and destroying cancer cells in the human body.
- Consideration
- DNA origami was very impressive and DNA origami technology development will continue.
So DNA origami can be adapted to create more complex or larger structures.